[本站讯]近日,控制科学与工程学院刘治平教授团队在国际学术期刊Advanced Science(中科院1区,IF=14.3)在线发表了题为“MuToN quantifies binding affinity changes upon protein mutations by geometric deep learning”的研究论文。本研究开发了一种名为MuToN的新型计算框架,通过几何深度学习模型精准量化了由氨基酸突变所引起的蛋白质结合亲和力的变化,这对理解复杂生物过程和疾病机制具有重要研究意义。博士研究生李朋湃为第一作者,刘治平教授为通讯作者,山东大学为第一作者单位和通讯作者单位。
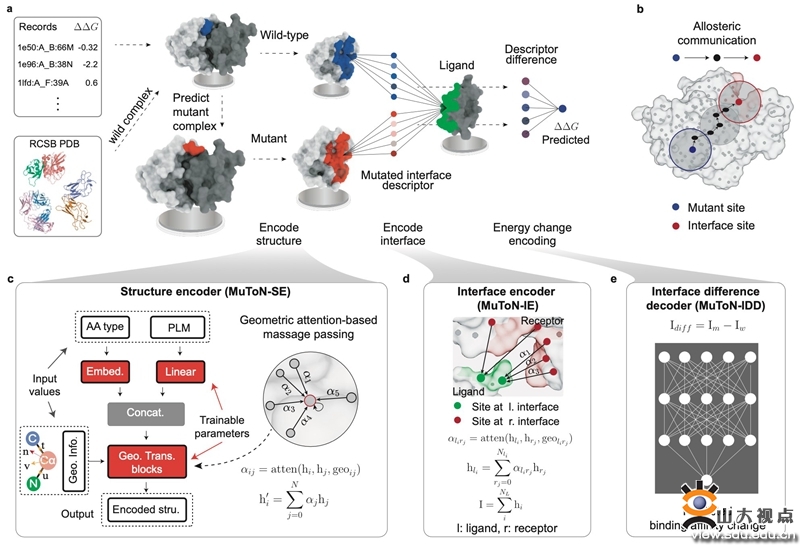
MuToN框架流程图
蛋白质在多种生物功能中发挥关键作用,通过与其他蛋白质和分子的相互作用来维系细胞活动的正常进行。由氨基酸序列变异所触发的突变效应,可能改变蛋白质与其他分子之间的结合模式,进而影响蛋白质的功能特性,最终导致疾病的发生。鉴于蛋白质结构的复杂性,包括其高维度及非欧几里得特性,如何精确量化这些变异对蛋白质结合亲和力模型的影响,已成为该领域的一大挑战。为应对该难题,MuToN框架充分利用了蛋白质几何表征的灵活性,设计了三个协同工作的子模块,全面模拟突变的生物学效应并量化其影响。首先是结构编码器(MuToN-SE)模块,该模块将野生型和突变型蛋白质三维结构编码为基于空间距离的残基级图,由此不仅捕捉了蛋白质突变所导致的局部空间环境变化,还揭示了潜在的变构通信(allosteric communication)效应。其次是界面编码器(MuToN-IE)模块,该模块专注于解析蛋白质复合物的结合界面,通过整合受体和配体两侧的信息,构建出能够反映蛋白质绑定界面特性的描述符。最后,界面差异解码器(MuToN-IDD)模块利用上述生成的界面描述符,计算野生型和突变复合物之间界面特性的差异,进而预测绑定亲和力的变化,从而实现了氨基酸突变效应的精确度量。
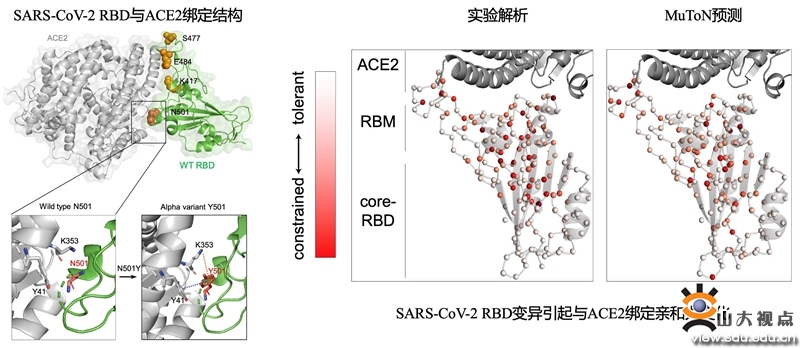
MuToN用于预测SARS-CoV-2突变引起的与ACE2绑定亲和力变化
本研究在多个基准数据集中验证了MuToN方法的有效性。实验结果表明,MuToN在预测结合自由能变化(ΔΔG)方面超越了已有方法,达到SOTA(state-of-the-art)水平。同时,MuToN方法具有高通量的优势,能够深入解析突变如何影响蛋白质与蛋白质之间的相互作用,进而预测生物过程的发展走向。例如,该方法能够准确预测病毒的变异如何改变其与宿主受体的结合特性,这对设计针对性的疫苗和治疗方法具有重要指导意义。作为具体的案例研究,本研究利用MuToN方法,准确预测出新型冠状病毒(SARS-CoV-2)刺突蛋白流行变体与关键宿主受体ACE2结合亲和力的动态变化趋势,精确绘制了SARS-CoV-2突变图谱,为提前识别新型冠状病毒的关键突变位点提供了可能性。
上述研究工作得到了国家重点研发计划、国家自然科学基金重大研究计划和山东大学集成攻关培育项目的大力支持。
原文链接:
https://onlinelibrary.wiley.com/doi/full/10.1002/advs.202402918
