[本站讯]近日,山东大学软件学院吴昊副教授团队在生成式人工智能研究方面取得新进展,相关成果以“scCross: a deep generative model for unifying single-cell multi-omics with seamless integration, cross-modal generation, and in silico exploration”为题发表在基因组学国际顶尖学术期刊Genome Biology。软件学院吴昊副教授和加拿大麦吉尔大学医学院丁俊研究员为论文通讯作者,软件学院硕士研究生杨秀辉为第一作者,山东大学软件学院为第一作者和通讯作者单位。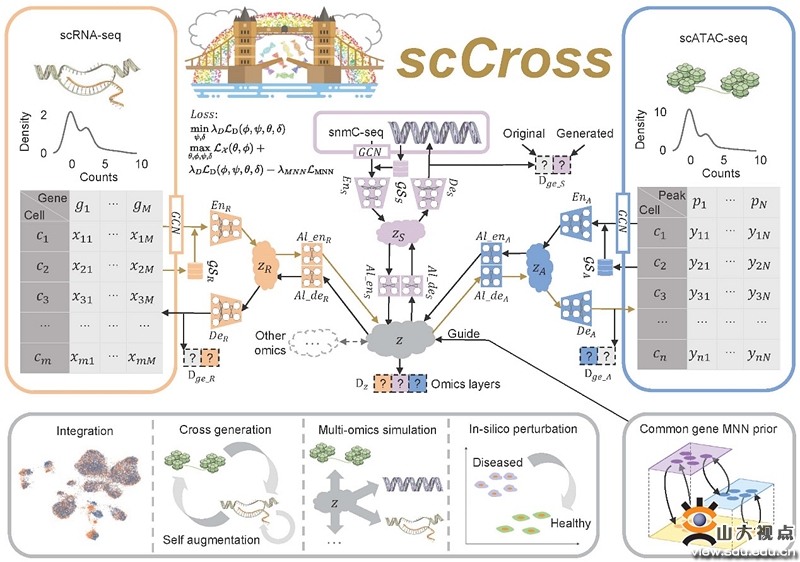
单细胞测序技术的引入使科学家能够以前所未有的精度解析细胞异质性。这一技术揭示了复杂的细胞动态,并对癌症生物学、神经生物学和药物发现等领域产生了重大影响。然而,这些技术生成的数据往往具有高度的复杂性和多样性,导致许多现有计算工具只能针对特定的数据模态提供片段化的视角。这种局限性限制了我们对细胞全景的全面理解。同时,不同模态数据存在数据量不平衡的特点。为应对这些挑战,吴昊副教授团队提出了生成式人工智能基础理论框架scCross集成模型。这一工具不仅在整合单细胞多组学数据方面表现出色,其最显著的特点是能够生成跨模态单细胞组学数据。这一能力使得研究人员能够在丰富数据模态与稀缺数据模态之间架起桥梁,从而实现更全面的细胞状态描绘。同时,scCross的另一重要功能是高保真模拟单细胞多组学数据,并且支持计算模拟扰动。这种能力使研究人员能够在数据整合的基础上进行细胞干预的虚拟实验,从而探索潜在的细胞干预策略。通过提供对跨模态细胞动态的深入理解,scCross不仅提升了单细胞多组学数据研究的实用性,也推动了该领域的创新与发展。
吴昊副教授团队长期致力于生成式人工智能、单细胞多组学数据集成、跨模态数据生成及下游分析和细胞生命周期预测、三维基因组结构及相关调控元件预测等相关研究,系列研究成果发表于Genome Biology ,Communications Biology,Briefings in Bioinformatics,Computers in Biology and Medicine,Neurocomputing等学术期刊,相关研究得到国家自然科学基金和国家重点研发计划等项目资助。
